Restrikční endonukleázy - Molekulární nůžky
Odůvodnění
Termín "restrikční enzym" pochází ze studií fága λ (fág lambda) Enterobacteria v laboratořích Wernera Arbera a Matthewa Meselsona. Byla studována schopnost některých E. coli kmenů inhibovat aktivitu fága lambda enzymatickým štěpením fágové DNA a enzym zodpovědný za tuto růstovou restrikci byl označen jako restrikční enzym.1, 2, 3
Werner Arber, Daniel Nathans a Hamilton O. Smithovi byla v roce 1978 udělena Nobelova cena za fyziologii nebo lékařství za objev a charakterizaci restrikčních enzymů, což vedlo k rozvoji technologie rekombinantní DNA.
Úvod
Restrikčním enzymům se také říká "molekulární nůžky", protože štěpí DNA v určitých rozpoznávacích sekvencích známých jako restrikční místa nebo v jejich blízkosti. Tyto enzymy provádějí jeden řez na každém ze dvou vláken DNA a nazývají se také restrikční endonukleázy.4
Viry infikují hostitelské buňky vpravením své DNA do buněk. Tato virová DNA unese mechanismus hostitelské buňky pro reprodukci virového potomstva, což má za následek smrt hostitelské buňky. K překonání virové infekce se u mnoha bakterií a archeí vyvinulo několik mechanismů. Hlavní ochranný mechanismus zahrnuje použití restrikčních enzymů k rozkladu napadené virové DNA jejím štěpením na specifických restrikčních místech. Hostitelská buňka zároveň chrání svou vlastní DNA před štěpením pomocí jiných enzymů zvaných metylázy, které metylují adeninové nebo cytosinové báze v rozpoznávacích sekvencích hostitele. Pro každý restrikční enzym produkuje hostitelská buňka odpovídající metylázu, která metyluje a chrání hostitelskou DNA před degradací. Tyto enzymy tvoří restrikčně-modifikační systémy (R-M).
Restrikční enzymy katalyzují hydrolýzu vazby mezi atomem 3'-kyslíku a atomem fosforu ve fosfodiesterové páteři DNA. Enzymy vyžadují ke své činnosti Mg2+ nebo jiné dvojmocné ionty.
Nomenklatura
Smith a Nathans navrhli v roce 1973 zásady pro pojmenování restrikčních endonukleáz. Podle těchto pokynů začínají názvy enzymů třípísmennou zkratkou psanou kurzívou. První písmeno označuje první písmeno bakteriálního rodu, ze kterého byl enzym izolován, a další dvě písmena jsou odvozena od bakteriálního druhu. Za nimi mohou následovat další písmena nebo číslice označující sérotyp nebo kmen. Následuje mezera a římská číslice, která označuje chronologii identifikace. Například, Hind III byl třetí ze čtyř enzymů izolovaných z Haemophilus influenza sérotypu d.6
Typy restrikčních enzymů
Podrobněji se věnujte této problematice.Na základě složení, vlastností štěpného místa a požadavků na kofaktory se restrikční endonukleázy dělí do čtyř skupin: typ I, II, III a IV.
Místo štěpení je v tabulce označeno červenou šipkou.
Faktory ovlivňující aktivitu restrikčních enzymů
V závislosti na substrátové DNA a reakčních podmínkách vykazují restrikční enzymy širokou variabilitu štěpné a případné hvězdicové aktivity. Pro dosažení požadovaného štěpení se stává důležitou kontrola následujících faktorů:
- Hvězdicová aktivita: Při neoptimálních reakčních podmínkách štěpí některé restrikční enzymy sekvence bází v místech odlišných od definované rozpoznávací sekvence. Jinými slovy, štěpí na nespecifických místech. Tento jev se nazývá hvězdicová aktivita. Mezi faktory, které vyvolávají hvězdicovou aktivitu, patří vysoká koncentrace soli a glycerolu, přítomnost nečistot, nadměrné množství enzymu v porovnání se substrátovou DNA, prodloužená doba inkubace nebo nekompatibilní pufr a kofaktor.
- Metylovaná DNA: Některé molekuly DNA jsou v rozpoznávacím místě metylované, což je činí odolnými vůči štěpení některými restrikčními enzymy. Například většina E. coli kmenů exprimuje metyltransferázy Dam nebo Dcm, které metylují specifická rozpoznávací místa za vzniku G6mATC, respektive C5mCA/TGG. G6mATC je odolný vůči štěpení pomocí Mbo I.
- Teplota: Většina endonukleáz optimálně štěpí cílovou DNA při 37 °C. Existují však výjimky s nižší nebo vyšší optimální teplotou. Například, Taq I optimálně tráví při 65 °C a Apa I (katalogové číslo 10899208001) tráví při 25 °C.
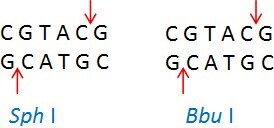
Isoschizomery a neoschizomery6
Isoschizomery jsou restrikční enzymy se stejnou rozpoznávací sekvencí a štěpnými místy. Příklad: Sph I (CGTAC/G) a Bbu I (CGTAC/G)
Neoschizomery jsou restrikční enzymy se stejnou rozpoznávací sekvencí, ale štěpí DNA v jiném místě této sekvence. Příklad: Tai I (ACGT/) a Mae II (A/CGT)
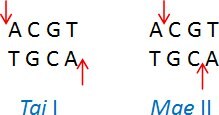
Produkty restrikčního štěpení
Restrikčním štěpením dvouřetězcové DNA vznikají dva druhy konců: lepivé konce a tupé konce.
Tupé konce mají 5'-fosfátovou skupinu, která podporuje ligaci. Jsou univerzálně kompatibilní s jinými DNA s tupými konci.
Tupé konce vytvářené EcoR V

Lepivé konce jsou malé úseky jednořetězcové DNA, které jsou schopné samovolné vazby nebo vazby s komplementární oblastí z jiné molekuly DNA. Lepivé konce mají 3'- nebo 5'-převisy o délce 1-4 nukleotidů.
5' kohezní konec generovaný Bln I (katalogové č. 1), který je určen k vytváření lepivých konců. 11558170001)

3' Soudržný konec generovaný Kpn I

Systém pufrů
Naše kolekce restrikčních enzymů byla optimalizována pro trávení pomocí pěti jedinečných pufrů. Při štěpení DNA pomocí jednoho enzymu používejte pufr dodávaný s enzymem. Při dvojitém štěpení DNA použijte pufr, ve kterém oba enzymy vykazují 100% aktivitu. Případně lze optimální pufr určit z tabulky běžných dvojitých štěpení. V některých případech se doporučuje sekvenční trávení z důvodu nekompatibility pufrů (složení nebo teplota). Protokoly restrikčního štěpení s jedním enzymem, dvěma restrikčními enzymy a sekvenčního štěpení DNA naleznete na Protokol štěpení restrikčními enzymy. Výběr správného pufru je rozhodující pro získání vysoké aktivity obou enzymů a pro zamezení hvězdicové aktivity. Výběr pěti pufrů umožňuje uživateli vybrat kompatibilní pufry pro požadovanou digesci. Většina digescí vyžaduje 1-2 pufry, což se stává nákladově efektivní.
Použití
Schopnost restrikčních endonukleáz štěpit DNA na specifických rozpoznávacích místech umožnila rozsáhlé využití těchto enzymů jako základních nástrojů v několika technikách molekulární biologie. Některé z hlavních aplikací jsou vysvětleny níže:
- Molekulární klonování: Oblíbeným použitím restrikčních enzymů je tvorba rekombinantních molekul DNA. Tento proces zahrnuje rozřezání donorové DNA (obvykle plazmidu) a vektorové DNA (obvykle genu z jiného organismu) restrikčním enzymem za účelem získání kompatibilních konců. Tyto konce mohou být buď "tupé", nebo "lepivé". Obě rozštěpené DNA se spojí dohromady pomocí enzymu zvaného DNA ligáza, čímž vznikne rekombinantní molekula DNA. Tuto rekombinantní DNA lze poté vnést do hostitelského organismu a replikovat ji. Podrobnější informace naleznete v Příručce pro klonování restrikčními enzymy
- Mapování DNA, známé také jako restrikční mapování, zahrnuje použití restrikčních endonukleáz k získání strukturních informací o fragmentu DNA nebo genomu. Mapování zahrnuje určení pořadí míst restrikčních enzymů v genomu. Zájmová DNA, jejíž struktura má být určena, se štěpí řadou restrikčních endonukleáz, čímž vznikají fragmenty DNA různé velikosti. Tyto fragmenty se rozdělí na agarózovém gelu, aby se určila struktura zájmové DNA.
Na základě známých míst restrikčních enzymů určitého fragmentu DNA lze restrikční endonukleázy použít k ověření identity tohoto fragmentu DNA. - Restrikční landmark genomické skenování je metoda analýzy genomu, která využívá kombinaci restrikčních enzymů k vizualizaci rozdílů v úrovni metylace napříč genomem daného organismu. Jedná se o užitečnou techniku k identifikaci odchylek od normálu v jakékoli DNA. Je velmi účinná při zjišťování hyper/hypometylace u nádorů, delecí nebo amplifikací genů nebo změn v expresi genů v průběhu vývoje organismu.8
- Sekvenování genů: Velkou molekulu DNA lze sekvenovat tak, že ji natrávíme restrikčními enzymy a výsledné fragmenty zpracujeme pomocí sekvenátoru DNA.
- Polymorfismus délky restrikčních fragmentů (RFLP) obsahuje trávení vzorku DNA pomocí restrikčních enzymů, oddělení těchto fragmentů na základě délky pomocí gelové elektroforézy a jejich přenesení na membránu. Tyto fragmenty se poté navážou na radioaktivní nebo fluorescenčně značenou sondu zaměřenou na specifické sekvence, které jsou v závorce v místech restrikčních enzymů. K RFLP dochází, když se výsledné délky fragmentů u jednotlivých jedinců liší. Každý jedinec má jedinečný vzor nazývaný "biologický čárový kód". Tato technika byla první technikou profilování DNA používanou při mapování genů, lokalizaci genů pro genetické poruchy, určování rizika onemocnění a testování otcovství.9
- Gelová elektroforéza v pulzním poli obsahuje separaci velkých fragmentů DNA, především fragmentů vzniklých štěpením bakteriálního genomu vzácným restrikčním enzymem. Vzniklý jedinečný vzor se používá k rozlišení různých kmenů bakterií. Může být užitečný k identifikaci konkrétního kmene jako příčiny rozšířeného onemocnění.10, 11
- Sériová analýza genové exprese (SAGE) je technika, která zahrnuje kvantitativní a simultánní analýzu velkého počtu transkriptů ve formě malých značek. Restrikční enzymy se v této technice používají jako kotvící enzym a značkovací enzym.12
- Integrace zprostředkovaná restrikčními enzymy (REMI) zahrnuje použití restrikčních enzymů k vytvoření kompatibilních kohezních konců v genomu pro vložení směsi plazmidové DNA, která byla linearizována restrikčním enzymem. Plasmidová DNA je transformována do hostitelské buňky spolu s restrikčním enzymem, který usnadňuje integraci DNA do kognitivních restrikčních míst v chromozomech. Tato technika je užitečná pro genetický screening a pro vkládání genetických a molekulárních markerů na určitá místa v genomu, aby bylo možné identifikovat zajímavé geny na základě jejich mutantních fenotypů.13
Materiály
Odkazy
Abyste mohli pokračovat ve čtení, přihlaste se nebo vytvořte účet.
Nemáte účet?