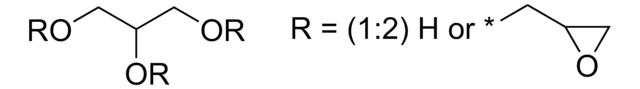SAB1400655
Anti-VISA antibody produced in mouse
IgG fraction of antiserum, buffered aqueous solution
Synonyme(s) :
Anti-CARDIF, Anti-DKFZp666M015, Anti-FLJ27482, Anti-FLJ41962, Anti-IPS-1, Anti-KIAA1271, Anti-MAVS
About This Item
Produits recommandés
Source biologique
mouse
Niveau de qualité
Conjugué
unconjugated
Forme d'anticorps
IgG fraction of antiserum
Type de produit anticorps
primary antibodies
Clone
polyclonal
Forme
buffered aqueous solution
Espèces réactives
human
Technique(s)
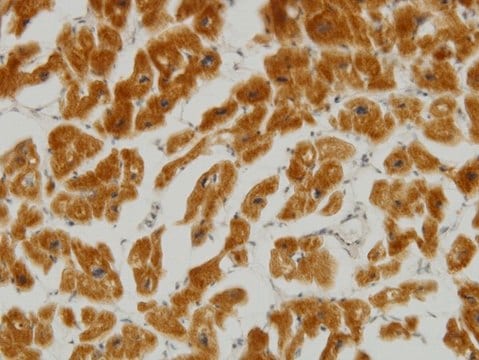
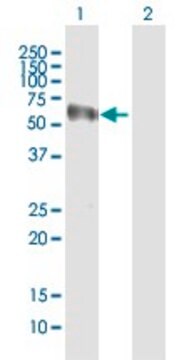
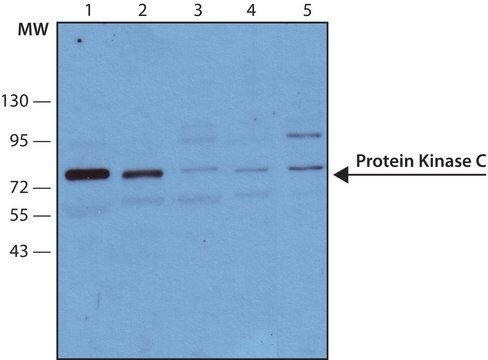
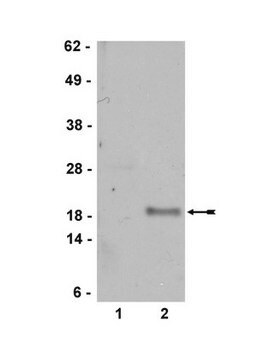
western blot: 1 μg/mL
Conditions d'expédition
dry ice
Température de stockage
−20°C
Modification post-traductionnelle de la cible
unmodified
Informations sur le gène
human ... VISA(57506)
Catégories apparentées
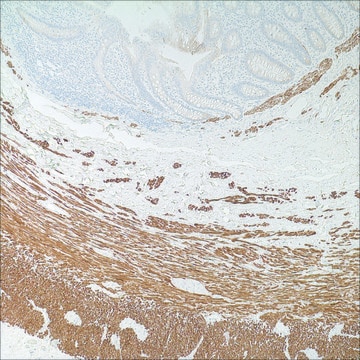
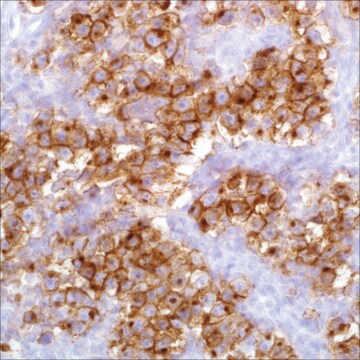
Description générale
Immunogène
Sequence
MPFAEDKTYKYICRNFSNFCNVDVVEILPYLPCLTARDQDRLRATCTLSGNRDTLWHLFNTLQRRPGWVEYFIAALRGCELVDLADEVASVYESYQPRTSDRPPDPLEPPSLPAERPGPPTPAAAHSIPYNSCREKEPSYPMPVQETQAPESPGENSEQALQTLSPRAIPRNPDGGPLESSSDLAALSPLTSSGHQEKDTELGSTHTAGATSSLTPSRGPVSPSVSFQPLARSTPRASRLPGPTGSVVSTGTSFSSSSPGLASAGAAEGKQGAESDQAEPIICSSGAEAPANSLPSKVPTTLMPVNTVALKVPANPASVSTVPSKLPTSSKPPGAVPSNALTNPAPSKLPINSTRAGMVPSKVPTSMVLTKVSASTVPTDGSSRNEETPAAPTPAGATGGSSAWLDSSFENRGLGSELSKPGVLASQVDSPFSGCFEDLAISASTSLGMGPCHGPEENEYKSEGTFGIHVAENPSIQLLEGNPGPPADPDGGPRPQADRKFQEREVPCHRPSPGALWLQVAVTGVLVVTLLVVLYRRRLH
Forme physique
Vous ne trouvez pas le bon produit ?
Essayez notre Outil de sélection de produits.
Code de la classe de stockage
10 - Combustible liquids
Point d'éclair (°F)
Not applicable
Point d'éclair (°C)
Not applicable
Faites votre choix parmi les versions les plus récentes :
Certificats d'analyse (COA)
Vous ne trouvez pas la bonne version ?
Si vous avez besoin d'une version particulière, vous pouvez rechercher un certificat spécifique par le numéro de lot.
Déjà en possession de ce produit ?
Retrouvez la documentation relative aux produits que vous avez récemment achetés dans la Bibliothèque de documents.
Notre équipe de scientifiques dispose d'une expérience dans tous les secteurs de la recherche, notamment en sciences de la vie, science des matériaux, synthèse chimique, chromatographie, analyse et dans de nombreux autres domaines..
Contacter notre Service technique