Spolehlivá extrakce DNA z karet Whatman® FTA®
Tato studie zkoumala výtěžnost a kvalitu DNA ze vzorků nanesených na karty Whatman® FTA® při použití pěti běžných metod extrakce DNA. Údaje ukazují, že všech pět metod poskytuje přečištěnou DNA v dostatečném množství a kvalitě pro kvantitativní PCR i analýzy krátkých tandemových repetic. Kromě toho se ukázalo, že zařízení EasiCollect™ pro odběr bukálních vzorků je kompatibilní se všemi pěti metodami extrakce DNA a poskytuje konečné výtěžky DNA stejné nebo vyšší než výtěžky ze samostatných pěnových tamponů.
Úvod
Skupina produktů Whatman® FTA® (obrázek 1) usnadňuje sběr, transport, purifikaci a dlouhodobé skladování nukleových kyselin při pokojové teplotě, a to vše v jediném zařízení. Technologie FTA má schopnost lyzovat buňky při kontaktu, denaturovat proteiny, odstraňovat kontaminanty a chránit DNA před degradací. V klasickém scénáři pro karty FTA se analyzuje přečištěná DNA, která je ještě vázána na matrici (tj. pomocí metody "punch-in"). Tato studie zkoumala alternativní možnost pro karty FTA - extrakci DNA do roztoku.
Materiály a metody
Sběr vzorků
Na karty FTA byla odebrána plná krev a bukální buňky.
Krev
Krev od deseti nepříbuzných jedinců byla odebrána venepunkcí do vakuových zkumavek obsahujících difosforečnan draselný EDTA. Krev (40 μl) byla nanesena na každou kartu FTA Micro Card. Karty byly sušeny nejméně 3 hodiny při pokojové teplotě.

Obrázek 1. Rodina produktů Whatman® FTA®.
Bukální vzorky
Bukální vzorky byly odebrány deseti nepříbuzným osobám pomocí zařízení EasiCollect od společnosti Cytiva (obrázek 2) nebo pěnových tamponů. Vzorky byly aplikovány na karty FTA podle pokynů výrobce. Karty byly sušeny po dobu nejméně 2 hodin při pokojové teplotě.

Obrázek 2. EasiCollect™ je univerzální zařízení, které odebírá bukální vzorky a přenáší je na integrovanou kartu FTA.
Metody extrakce DNA
DNA byla extrahována nebo izolována z karet FTA pomocí pěti různých metod, přesně podle popisu v následujících protokolech. Mezi tyto metody patří např:
- Organická extrakce
- Pryskyřice Chelex 100 (142-1253, Bio-Rad Laboratories,vHercules, CA, USA)
- Sada QIAamp™ DNA Investigator Kit (56504, Qiagen, Valencia, CA, USA)
- illustra™ tissue and cells genomicPrep Mini Spin Kit (Cytiva)
- DNA IQ™ Kit (TB297, Promega Corp, Madison, WI, USA)
Organická extrakce DNA
Připravte si extrakční pufr a zásobní roztok Proteinázy K. Extrakční pufr: 10 mM Tris-HCl, pH 8,0; 10 mM EDTA, disodná sůl, pH 8,0; 100 mM chlorid sodný; a 2% v/v SDS. Zásobní roztok proteinázy K: 20 mg/ml ve sterilní destilované vodě.
- Pomocí jednorázového děrovače Harris Uni-Core (1,2, 2,0, 3,0 nebo 6,0 mm) odeberte disk ze středu vysušeného místa vzorku. Vložte disk do čisté 1,5 ml mikrocentrifugační zkumavky bez RNázy/DNázy.
- Přidejte 500 μl extrakčního pufru a 20 μl základního roztoku Proteinázy K.
- Vortexujte a inkubujte při 56 °C přes noc za míchání.
- Přidejte stejný objem pufrovaného fenolu (pH 8,0), krátce vortexujte a odstřeďujte 10 min při maximální rychlosti v mikrocentrifuze.
- Přeneste horní vodnou fázi do nové mikrocentrifugační zkumavky, která obsahuje 500 μl chloroformu.
- Důkladně promíchejte a odstřeďujte 10 min při maximální rychlosti v mikrocentrifuze.
- Převeďte horní, vodnou fázi do nové zkumavky obsahující 50 μl 3 M octanu sodného o pH 5,2.
- Přidejte 800 μl 100% ethanolu, vortexujte a srážejte při -20 °C po dobu nejméně 1. hodiny.5 h.
- Znovu získáte DNA odstřeďováním po dobu 30 min při maximálních otáčkách v mikrocentrifuze a dekantací supernatantu.
- Přidejte 1 ml 70% ethanolu k peletě a odstřeďujte 20 min při maximálních otáčkách v mikrocentrifuze.
- Peletku sušte 30 min ve vakuové centrifuze.
- Peletku rozpusťte v 50 μl TE pufru nebo vody.
Izolace DNA pomocí pryskyřice Chelex 100
Před použitím připravte 5% w/v suspenzi pryskyřice Chelex 100 ve sterilní vodě; míchejte, aby se suspenze udržela.
- Pomocí jednorázového děrovače Harris Uni-Core (1,2, 2,0, 3,0 nebo 6,0 mm) odeberte kotouček vzorku ze středu vysušeného místa vzorku. Umístěte disk do čisté 1,5 ml mikrocentrifugační zkumavky bez RNázy/DNázy.
- Macerujte disk FTA jehlou o průměru 20 mm.
- Promyjte disk přidáním 1 ml sterilní vody do každé zkumavky se vzorkem a inkubujte při pokojové teplotě po dobu 10 min za občasného víření. Odstraňte vodu.
- Proplachování opakujte přidáním 1 ml sterilní vody do každé zkumavky se vzorkem a inkubujte při pokojové teplotě po dobu 10 min za občasného víření.
- Centrifugujte vzorky po dobu 3 min při 20 000 x g.
- Odstraňte a zlikvidujte supernatant.
- Přidejte 200 μl suspenze Chelex 100 pomocí pipetovací špičky s velkým otvorem.
- Inkubujte vzorek při 56 °C po dobu 20 min.
- Přibližně 15 s vířivě promíchávejte.
- Inkubujte při 100 °C po dobu 8 min.
- Přibližně 15 s vířivě promíchávejte.
- Centrifugujte vzorek po dobu 3 min při 20 000 x g.
- Přeneste supernatant do nové sterilní předem označené mikrocentrifugační zkumavky, přičemž dávejte pozor, abyste nepřenesli pelet pryskyřice Chelex 100.
- Krátkodobě uchovávejte DNA při teplotě 2 °C -8 °C nebo dlouhodobě při -20 °C.
Izolace DNA pomocí soupravy QIAamp DNA Investigator Kit
Souprava obsahuje reagencie a kolonky.
- Pomocí jednorázového děrovače Harris Uni-Core (1,2, 2,0, 3,0 nebo 6,0 mm) odeberte kotouček vzorku ze středu vysušeného místa vzorku. Umístěte disk do čisté 1,5 ml mikrocentrifugační zkumavky bez RNázy/DNázy.
- Přidejte do zkumavky 280 μl ATL pufru.
- Přidejte 20 μl Proteinázy K a 30 s vortexujte.
- Zahřívejte 60 min při 56 °C a každých 10 min 30 s vortexujte.
- Centrifugujte při maximální rychlosti v mikrocentrifuze po dobu 30 s.
- Přidejte 300 μl AL pufru a vortexujte po dobu 10 s.
- Inkubujte při 70 °C po dobu 10 min, každé 3 min vortexujte po dobu 10 s.
- Centrifugujte při maximální rychlosti po dobu 30 s a přeneste lyzát do minElute kolony.
- Centrifugujte při 6000 x g po dobu 1 min a zlikvidujte průtok.
- Přidejte 700 μl pufru AW2 a centrifugujte po dobu 1 min při 6000 x g. Zlikvidujte průtok.
- Přidejte 700 μl 100% ethanolu a centrifugujte po dobu 1 min při 6000 x g. Průtok zlikvidujte.
- Centrifugujte při 20 000 x g po dobu 3 min, poté otevřete víko kolony na 10 min při pokojové teplotě.
- Přidejte 50 μl destilované vody a inkubujte 5 min při pokojové teplotě.
- Centrifugujte při 20 000 x g po dobu 1 min. Průtok, který obsahuje extrahovanou DNA, uchovávejte při teplotě 4 °C, dokud nebude potřeba.
Izolace DNA pomocí soupravy illustra tissue and cells genomicPrep Mini Spin Kit
Souprava obsahuje činidla a kolonky. Potřebný je také fyziologický roztok pufrovaný fosfáty (PBS).
- Pomocí jednorázového děrovače Harris Uni-Core (1,2, 2,0, 3,0 nebo 6,0 mm) odeberte kotouček vzorku ze středu vysušeného místa vzorku. Umístěte disk do čisté 1,5 ml mikrocentrifugační zkumavky bez RNázy/DNázy.
- Přidejte 1 ml PBS a centrifugujte při 16 000 x g.
- Macerujte disk pomocí injekční jehly 20.
- Centrifugujte 10 s při 2000 x g.
- Přidejte ke každému vzorku 50 μl pufru 1.
- Přidejte ke každému vzorku 10 μl proteinázy K a 15 s vortexujte.
- Inkubujte 1 h při 56 °C
- Centrifugujte 10 s při 2000 x g. Přidejte 5 μl RNázy A (20 mg/ml). Inkubujte 15 min při pokojové teplotě.
- Přidejte 500 μl pufru 4 a 15 s vortexujte.
- Inkubujte 10 min při pokojové teplotě.
- Pipetujte vzorek do minikolonky (umístěné ve sběrné zkumavce) a odstřeďujte 1 min při 11 000 x g. Průtok zlikvidujte.
- Přidejte 500 μl pufru 4 do každé kolonky. Odstřeďujte 1 min při 11 000 x g a průtok zlikvidujte.
- Vložte kolonku do sběrné zkumavky a přidejte do ní 500 μl pufru 6.
- Centrifugujte 3 min při 11 000 x g. Přeneste kolonu do nové 1,5 ml zkumavky.
- Přidejte ke koloně 50 μl předehřátého elučního pufru (pufr 6).
- Inkubujte 1 min při pokojové teplotě.
- Centrifugujte 1 min při 11 000 x g. Vyhoďte kolonku a uchovávejte DNA při 4 °C, dokud nebude potřeba.
Izolace DNA pomocí soupravy DNA IQ
Souprava obsahuje činidla a kolonky. Potřebný je také magnetický separační stojan.
- Pomocí jednorázového děrovače Harris Uni-Core (1,2, 2,0, 3,0 nebo 6,0 mm) odeberte kotouček vzorku ze středu vysušeného místa vzorku. Umístěte disk do čisté 1,5 ml mikrocentrifugační zkumavky bez RNázy/DNázy.
- Přidejte 100 μl lyzačního pufru a inkubujte 30 min při 70 °C.
- Přeneste obsah do odstředivé kolony usazené v 1,5 ml zkumavce a odstřeďujte 2 min při maximální rychlosti. Odvzdušňovací kolonku zlikvidujte.
- Vyrobenou pryskyřici důkladně promíchejte a přidejte 7 μl pryskyřice pro 1,2 mm disk, 7 μl pro 2,0 mm disk, 16 μl pro 3,0 mm disk nebo 28 μl pro 6,0 mm disk.
- Vortexujte zkumavky po dobu 3 s a inkubujte při pokojové teplotě po dobu 5 min, přičemž jednou za minutu vortexujte po dobu 3 s.
- Vortexujte zkumavku po dobu 2 s a umístěte ji na magnetický stojan.
- Dekantujte supernatant, aniž byste narušili pelet.
- Přidejte 100 μl lyzačního pufru. Odstraňte zkumavku z magnetického stojanu a 2 s vortexujte.
- Vraťte zkumavku na magnetický stojan a dekantujte lyzační pufr.
- Přidejte 100 μl 1× promývacího pufru. Vyjměte zkumavku ze stojanu a 2 s vortexujte.
- Vraťte zkumavku do magnetického stojanu a dekantujte promývací pufr.
- Zopakujte promývací kroky 10 a 11 ještě dvakrát pro celkem tři promývání. Po posledním promývání dekantujte promývací pufr.
- Se zkumavkou v magnetickém stojanu otevřete víko a nechte ji 10 min vysychat na vzduchu.
- Přidejte 100 μl elučního pufru. Zavřete víko a 2 min vortexujte. Inkubujte 5 min při 65 °C.
- Vyjměte zkumavku z tepla a 2 s vortexujte, poté zkumavku okamžitě umístěte do magnetického stojanu.
- Převeďte eluát do čerstvé zkumavky a uchovávejte eluovanou DNA při 4 °C, dokud nebude potřeba.
Analýzy DNA
DNA extrahovaná z karet FTA byla měřena pomocí kvantitativní PCR (qPCR). DNA byla podrobena analýze krátkých tandemových repetic (STR) pro posouzení její kvality.
Kvantifikace DNA pomocí qPCR
Extrahovaná DNA byla měřena pomocí kvantitativní PCR (qPCR) a systému ABI™ 7900HT Fast Real-Time PCR System (Applied Biosystems, Foster City, CA, USA). Reakce byly připraveny pomocí sady Quantifiler™ Human DNA Quantification Kit (4343895, Applied Biosystems). Test kvantifikace DNA kombinuje dva 5' nukleázové testy. První je test specifický pro lidskou DNA, který se skládá ze dvou primerů pro amplifikaci lidské DNA a jedné sondy TaqMan™ MGB značené barvivem FAM™ pro detekci amplifikované sekvence. Druhým testem je test vnitřní kontroly PCR (IPC), který se skládá ze dvou primerů pro amplifikaci templátové DNA IPC a jedné sondy TaqMan MGB značené barvivem VIC™ pro detekci amplifikované DNA IPC. Standardní křivky pro kvantifikaci DNA byly připraveny pomocí kontrolní DNA dodané se sadou.
Analýza krátkých tandemových repetic
STR analýza extrahované DNA byla provedena pomocí systému Powerplex™ 16 (Promega) podle pokynů výrobce. Amplifikace byla provedena v termálním cykleru 9700 (Applied Biosystems) a produkty amplifikace byly analyzovány na genetickém analyzátoru ABI PRISM™ 3130XL (Applied Biosystems) podle pokynů výrobce.
Výsledky a diskuse
Kvantifikace DNA pomocí qPCR
DNA byla extrahována z disků FTA obsahujících krev nebo buňky bukální sliznice od deseti nepříbuzných jedinců. Pro každou extrakční metodu byly použity čtyři velikosti disků a celkové množství extrahované DNA na jeden disk bylo stanoveno podle popisu v části Materiály a metody.
Pro každou z pěti extrakčních metod bylo ze vzorků krve na kartách FTA získáno minimálně 10 ng genomové DNA (obr. 3). Množství DNA extrahované každou metodou se zvyšovalo s rostoucí velikostí disku.
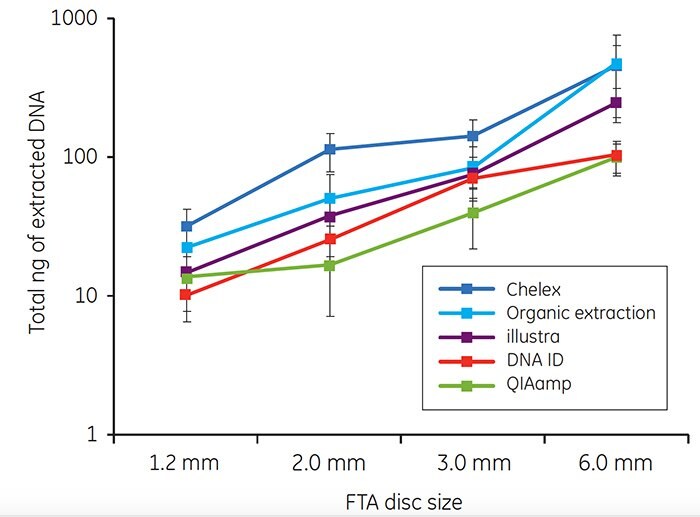
Obrázek 3. Celková DNA (ng) izolovaná z disků odebraných ze středu karet FTA, na které bylo naneseno 40 μl plné lidské krve. Každý bod na grafu představuje průměrnou výtěžnost DNA z jednoho disku od deseti jedinců. Chybové úsečky představují směrodatné odchylky. Všimněte si, že osa y představuje logaritmickou stupnici.
Výtěžnost vzorků DNA z bukální sliznice byla v minulosti variabilnější než u vzorků krve. Variabilita ve výtěžnosti bukální DNA je s největší pravděpodobností způsobena shlukováním buněk na matrici. Stejně jako u krevních vzorků mělo množství DNA získané jednotlivými extrakčními metodami tendenci se zvyšovat s rostoucí velikostí disku. Všimněte si, že vyšší výtěžnost DNA byla často získána z bukálních vzorků odebraných pomocí zařízení EasiCollect než ze vzorků odebraných pomocí pěnových tamponů (obrázek 4). Údaje uvedené na obrázcích 3 a 4 ilustrují, že běžně používané metody a soupravy pro extrakci DNA poskytují DNA v dostatečném množství a kvalitě pro úspěšnou qPCR.
DNA extrahovaná z odebraných bukálních vzorků

Obrázek 4. Celková DNA (ng) izolovaná z disků odebraných ze středu karty FTA obsahující buňky bukální sliznice, nanesené buď pěnovým aplikátorem, nebo zařízením EasiCollect, jak je uvedeno,
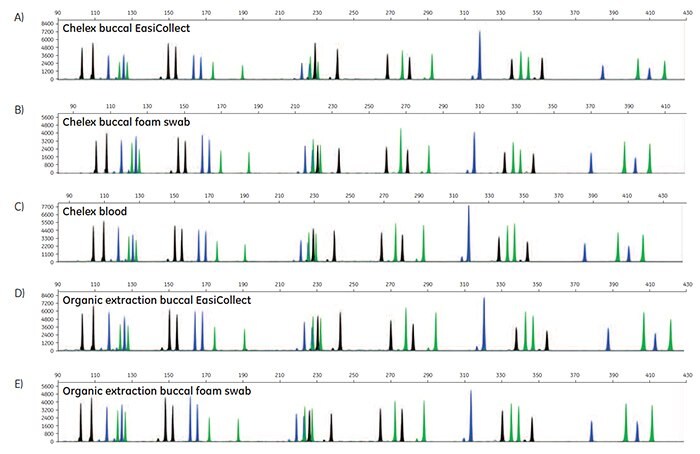
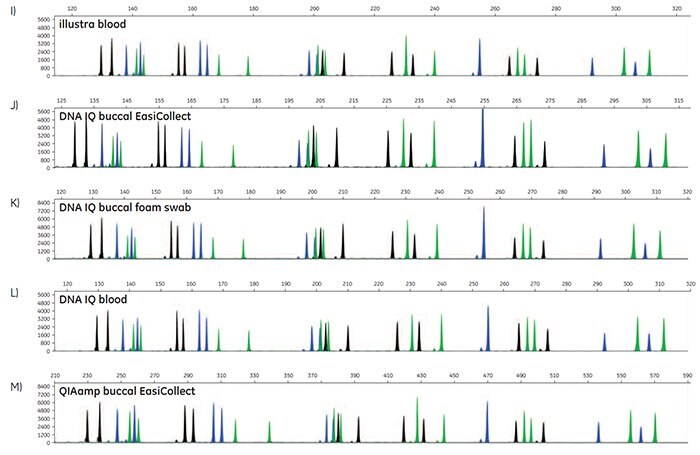
Obrázek 5. A-O. STR profily DNA extrahované z karet FTA obsahujících krevní nebo bukální vzorky testovaného subjektu 1.
STR data byla generována za účelem stanovení přesnosti volání alel pro DNA extrahovanou z karet FTA. Pro každou metodu extrakce DNA byly provedeny duplikáty DNA z karet FTA pro tři vzorky krve, tři vzorky bukální sliznice odebrané pomocí zařízení EasiCollect a tři vzorky bukální sliznice odebrané pomocí samostatných pěnových tamponů, což dává celkem 18 vzorků pro každou metodu extrakce DNA. Všimněte si, že tyto tři vzorky pocházely od tří testovaných osob. Z 90 odebraných profilů STR poskytlo 87 vzorků kvalitní údaje s přesným voláním alel na první pokus; tři vzorky vyžadovaly opakovanou injekci pro 100% přesné volání alel. Tyto údaje ilustrují, že běžně používané metody a soupravy pro extrakci DNA poskytují DNA v dostatečném množství a kvalitě pro podporu 100% přesnosti volání alel při analýze STR.
Závěry
Tato studie ukazuje, že pět běžných metod extrakce DNA poskytlo dostatek DNA z karet FTA pro následnou kvantitativní analýzu. Tato DNA byla dostatečně kvalitní pro určení lidské identity pomocí STR profilů. Kromě toho odběr bukálních buněk pomocí zařízení EasiCollect často vedl k vyšší výtěžnosti DNA než odběr pomocí pěnového tamponu. Tyto údaje ukazují, že zařízení EasiCollect je kompatibilní se současnými ověřenými laboratorními protokoly pro extrakci DNA.
Materiály
Odkazy
Abyste mohli pokračovat ve čtení, přihlaste se nebo vytvořte účet.
Nemáte účet?